itmo_conspects
Лекция 4. Нелинейные методы уменьшения размерности
Стохастическое вложение соседей с t-распределением
Стохастическое вложение соседей с t-распределением (t-distributed Stochastic Neighbor Embedding, t-SNE) - алгоритм, хорошо подходящий для визуализации данных в низкой размерности
Такой метод моделирует данные так, что близлежащие точки после алгоритма находятся рядом, а далеко стоящие с высокой вероятностью будут далеко друг от друга
Для этого:
-
Для каждой пары точек $x_i$ и $x_j$ вычисляется евклидово расстояние $d_{ij} = |x_i - x_j|$
-
Далее определяется вероятность того, что точка $x_j$ будет соседом точки $x_i$:
\[p_{j|i} = \frac{e^{-\frac{d^2_{ij}}{2\sigma_i^2}}}{\sum_{k \neq i} e^{-\frac{d^2_{ik}}{2\sigma^2_i}}}\]То есть доля $F_\xi (d_{ij})$ от суммы $\sum_{k \neq i} F_\xi (d_{ik})$ для всех точек, где $\xi$ - случайная величина из $N(0, \sigma_i^2)$
-
Для метода задается параметр перплексии $\mathrm{Perp}$. От него определяется значение $\sigma_i$ такое, что $\mathrm{Perp}(P_i) = 2^{H(P_i)}$, где $H(P_i) = - \sum_{j\neq i} p_{j|i} \log_2 p_{j|i}$ - энтропия Шеннона
В t-SNE функция перплексии $\mathrm{Perp}(P_i)$ устанавливается на какое-то число (оно называется
perplexity, обычно от 1 до 100), благодаря которому можно вывести $\sigma_i$Чем больше перплексия, тем больше вероятность того, что некоторая точка будет соседом для другой точки
При этом перплексия не должна быть больше $n - 1$, где $n$ - размер датасета
-
Совместная вероятность $p_{ij}$ определяется как $p_{ij} = \frac{p_{j|i} + p_{i|j}}{2n}$, при этом $p_{ii} = 0$
Заметим, что $p_{j|i} \neq p_{i|j}$
-
Пусть точки $y_i$ и $y_j$ - отображения точек $x_i$ и $x_j$ на целевом пространстве низкой размерности. Тогда установим, что вероятность того, что $y_i$ и $y_j$ - соседи, равна
\[q_{ij} = \frac{(1 + \|y_i - y_j\|^2)^{-1}}{\sum_{k \neq l} (1 + \|y_k - y_l\|^2)^{-1}}\]При этом $q_{ii} = 0$
Здесь берется функция плотности $F_t = \frac{\Gamma\left(\frac{n + 1}{2}\right) \left(1 + \frac{x^2}{n}\right)^{-\frac{n + 1}{2}}}{\sqrt{n\pi} \Gamma\left(\frac{n}{2}\right)}$ случайной величины $t$ из распределения Стьюдента $T_n$ при степени свободы $n = 1$, тогда
\[q_{ij} = \frac{F_t(\|y_i - y_j\|)}{\sum_{k \neq l} F_t(\|y_k - y_l\|)}\] -
Если вы дочитали до этого момента, то тут берется функция расстояния Кульбака-Лейблера (или сумма дивергенций Кульбака-Лейблера)
\[\mathrm{KL}(P \| Q) = \sum_{i \neq j} p_{ij} \log \frac{p_{ij}}{q_{ij}},\]которая с помощью градиентного спуска (метод нахождения минимума многомерной функции) минимизируется по отношению к точкам $y_i$, которые изначально были установлены случайным образом (отсюда стохастический)
Точки $y_i$ в ходе градиентного спуска “притягиваются” к своим местам
Количеством итераций алгоритма t-SNE называется количество шагов градиентного спуска - чем больше, тем точнее. На больших датасетах берется 500-2000, для приблизительной быстрой оценки 250-500
Наконец-то, мы получили точки $y_i$, которые можно отобразить на плоскости
Распределение Стьюдента с одной степенью свободы имеет более тяжелые хвосты, чем нормальное распределение. Это позволяет близким точкам оставаться очень близкими,а далеким точкам быть очень далекими
Алгоритмическая сложность вычисляется так:
- Вычисление попарных расстояний - $O(n^2 d)$
- Поиск $\sigma_i$ для каждой точки - $O(n^2 \log \mathrm{Perp})$
- Каждая итерация градиентного спуска - $O(n^2 k)$
где $d$ - размерность исходного пространства, $k$ - размерность целевого
Пример: есть датасет с 15 фруктами (яблоки и цитрусы), для них мы знаем кислотность, сладость и сочность
Алгоритм t-SNE с perplexity=4 явно отделит их и расположит на плоскости:
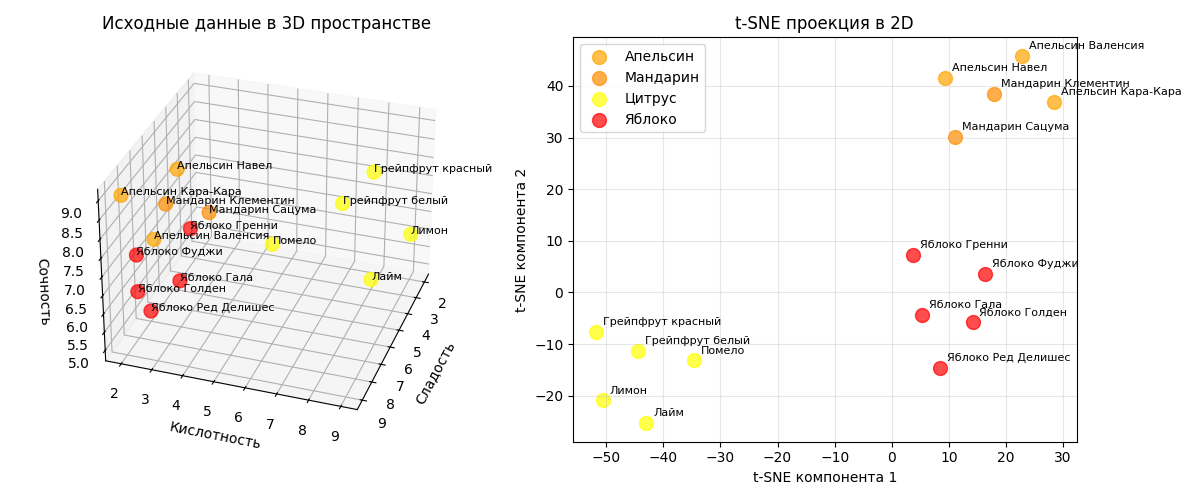
При этом также явно можно заметить, что на проекции они кластеризовались по своим признакам
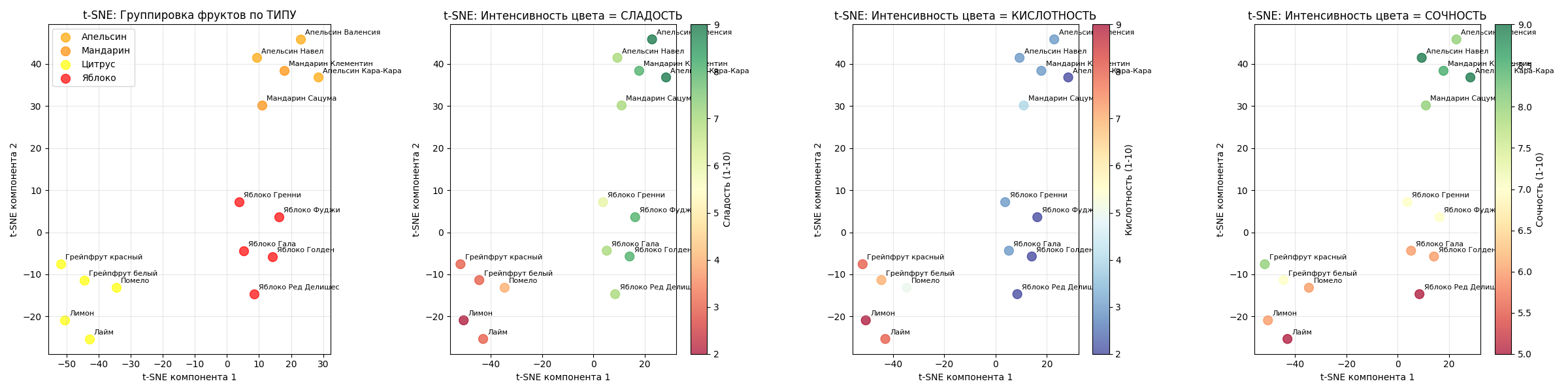
Код примера - machlearn_tsne_example.py
Другой пример - есть датасет с изображениями цифр от 0 до 9. Изображение состоит из сетки 8 на 8 (256 пикселей), где один пиксель - число от 0 до 1, обозначающий оттенок серого
Тогда можно понаблюдать, что происходит при разных perplexity:
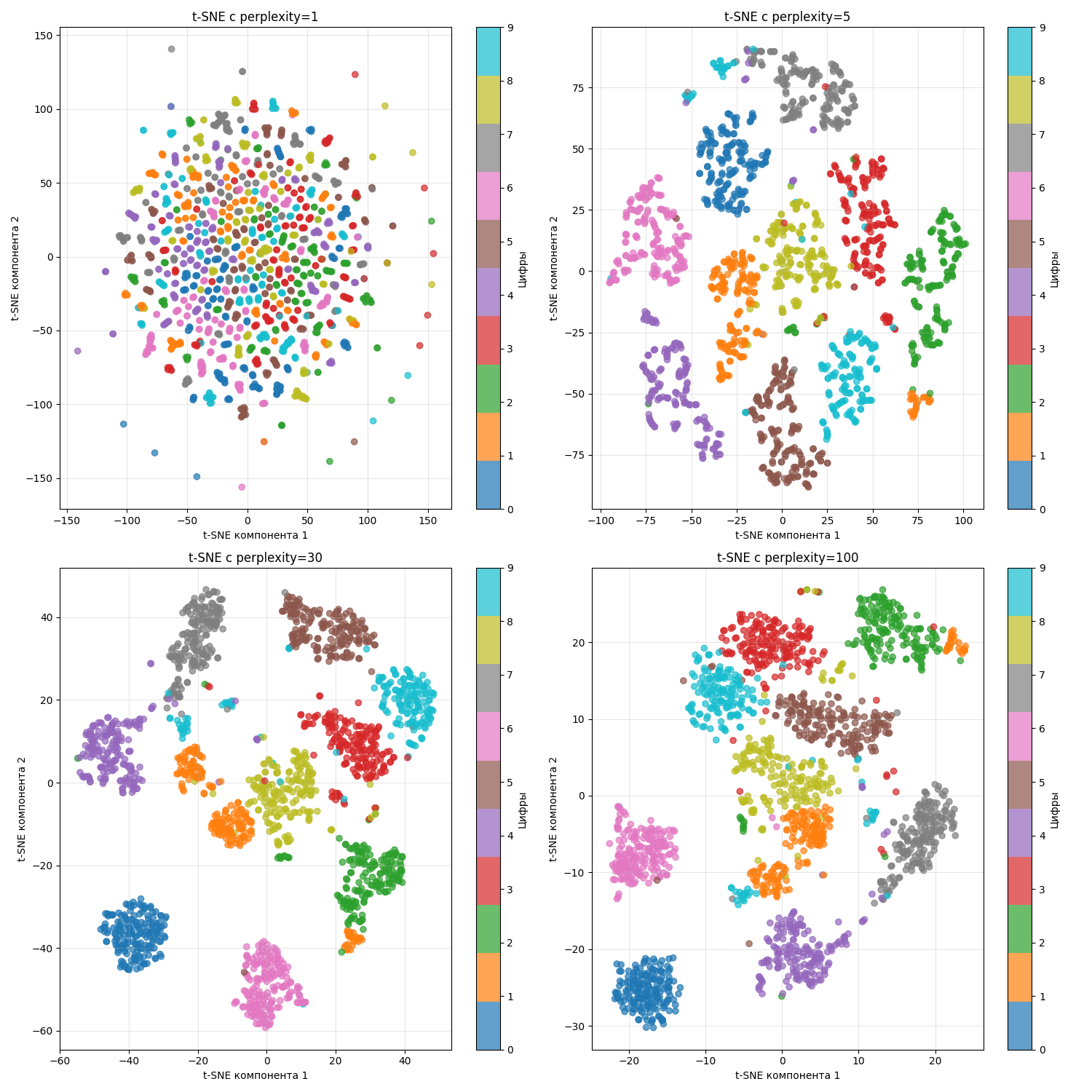
При маленьком perplexity образуются маленькие кластеры, а при большом - они “слипаются”
Метод t-SNE используется для:
- Для визуализации
- Для кластеризации
Однако надо учитывать его недостатки:
- t-SNE вычислительно дорогой
- Не сохраняет глобальную структуру
- Чувствительный к значению
perplexity
Алгоритм UMAP
Алгоритм UMAP (Uniform Manifold Approximation and Projection) - алгоритм, похожий на t-SNE. Алгоритм UMAP был создан в 2018 году (статья - *тык*) с целью получить более сильное математическое обоснование
Работает он так:
-
Даны параметры $k = \text{n\_neighbors}$ - заданное число ближайших соседей у точки и $\text{min\_dist}$ - минимальное расстояние между точками в целевом пространстве
-
Далее для каждое точки $x_i$ ищется $k$ ближайших соседей $T = {t_1, \dots, t_k}$, используя в качестве расстояния любимую метрику $\mathrm{dist}(x_i, t_i)$ (например, евклидово расстояние \(\mathrm{dist}(x_i, t_i) = \|x_i - t_i\|\))
-
Теперь для каждой точки вычисляет расстояние до самого ближнего соседа $\displaystyle \rho_i = \min_{t \in T} \mathrm{dist}(x_i, t)$
Также вычисляется $\sigma_i$ из уравнения $\displaystyle \sum_{t \in T} e^{-\frac{\mathrm{dist}(x_i, t) - \rho_i}{\sigma_i}} = \log_2 k$
-
Теперь строится взвешенный ориентированный граф, где вес ребра из точки $x_i$ в точку $x_j$ определяется как $v(x_i \to x_j) = e^{-\frac{\mathrm{dist}(x_i, x_j) - \rho_i}{\sigma_i}}$
-
Этот граф превращается в взвешенный неориентированный, тогда вес ребра из точки $x_i$ в точку $x_j$ определяется как $v_{ij} = v(x_i \to x_j) + v(x_j \to x_i) - v(x_i \to x_j) \cdot v(x_j \to x_i)$
-
После этого случайным образом создается новый граф в целевом пространстве меньшей размерности, с тем же количеством вершин, ребер и соответственными степенями вершин. В нем вес ребра считается как
\[w_{ij} = \frac{1}{1 + a \cdot \mathrm{dist}(y_i, y_j)^{2b}}\]Параметры $a$ и $b$ подбираются так, что бы $\frac{1}{1 + a \cdot \text{min \_ dist}^{2b}} = 0.5$, а значение производной $\frac{\partial w_{ij}}{\partial \mathrm{dist} (y_i, y_j)} = -1$ в точке $\mathrm{dist}(y_i, y_j) = \text{min \_ dist}$
-
Теперь составляется функция расстояний Кульбака-Лейбнера
\[\mathrm{KL}(P \| Q) = \sum_{i \neq j} v(x_i, x_j) \log \frac{v(x_i, x_j)}{w(y_i, y_j)} + (1 - v(x_i, x_j)) \log \left(\frac{1 - v(x_i, x_j)}{1 - w(x_i, x_j)}\right),\]которая с помощью стохастического градиентного спуска минимизируется
Алгоритм градиентного спуска работает фиксированное число итераций (так называемых эпох)
Теперь мы получаем координаты точек $y_i$ в пространстве меньшей размерности
Алгоритмическая сложность UMAP вычисляется так:
- Нахождение ближайших соседей - $O(n^2)$ (с оптимизациями можно $O(n \log n)$)
- Построение графа - $O(nk)$
- Градиентный спуск - $O(Tnk)$, где $T$ - количество эпох
Получается $O(n \log n + Tnk)$
Алгоритм UMAP выходит быстрее на больших выборках данных (при $n > T$)
Пример: возьмем этот же датасет с 15 фруктами (яблоки и цитрусы). Алгоритм UMAP с n_neighbors=5 и min_dist=0.1 отделит фрукты с разными параметрами
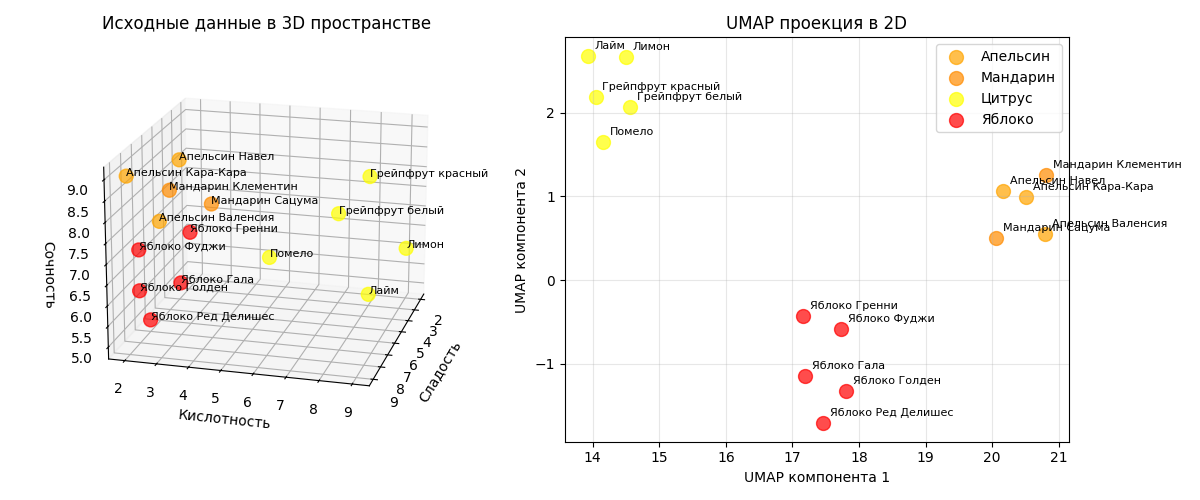
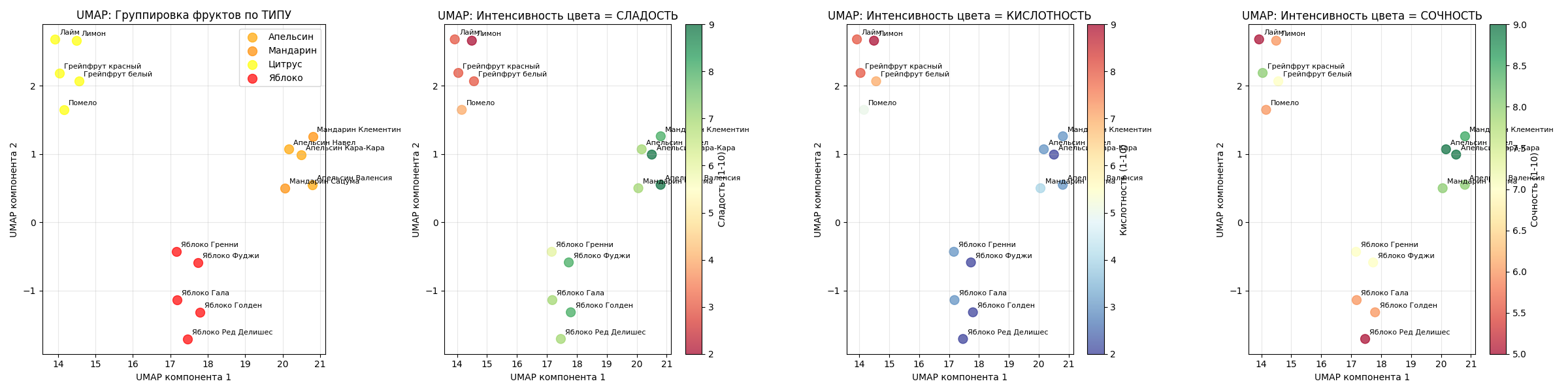
На маленьких датасетах, как можно заметить, при правильно подобранных параметрах результат UMAP мало отличим от t-SNE
Код примера - machlearn_umap_example.py
Посмотрим, что происходит при разных n_neighbors и min_dist на датасете с изображениями цифр:
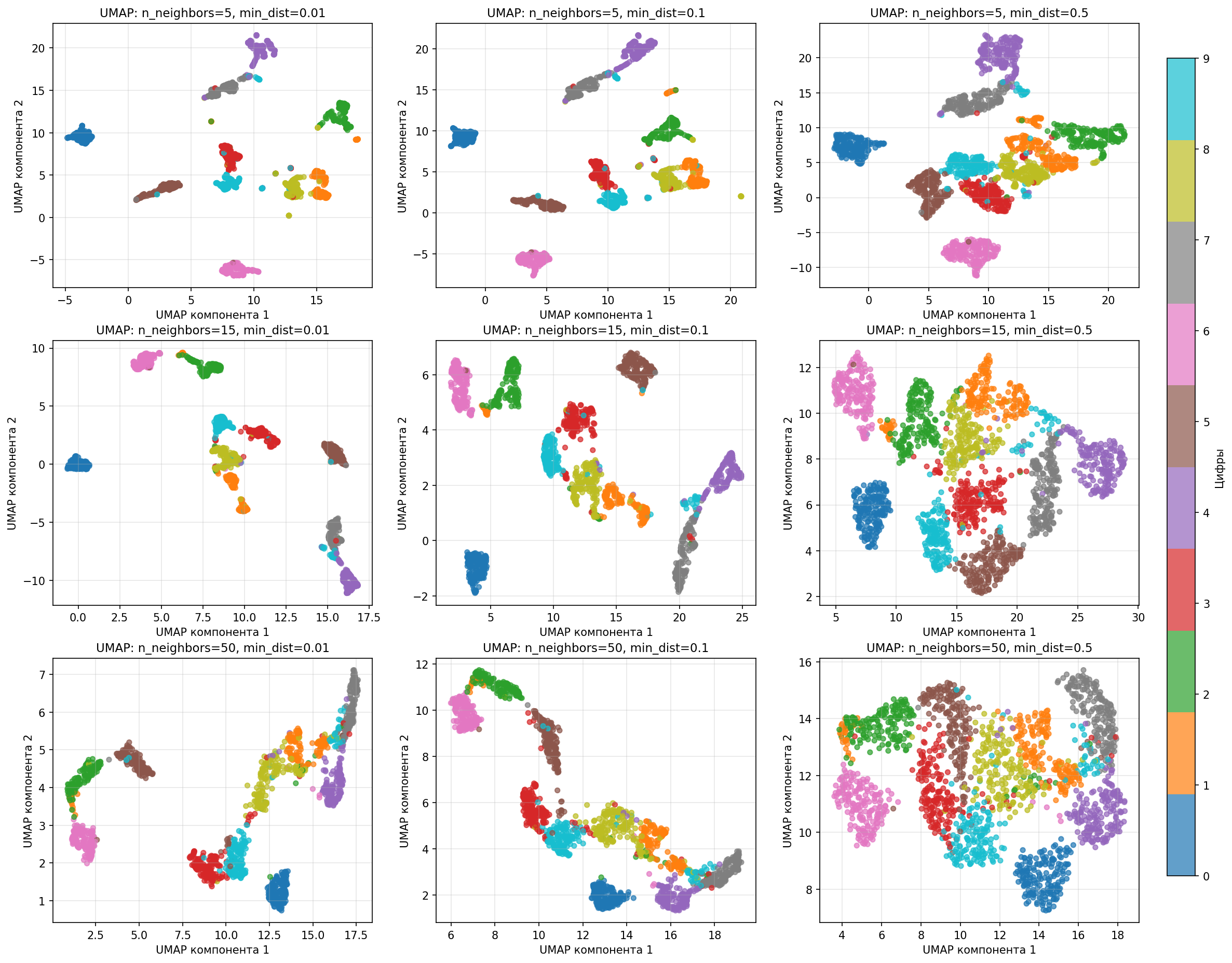
Малое значение n_neighbors подчеркивает локальную структуру, а большое - связи между кластерами, то есть n_neighbors влияет на масштаб. Расстояние min_dist влияет на плотность кластера на графика
На практике параметры n_neighbors и min_dist определяются методом тыка, но хорошими начальными значениями являются $\text{n\_neighbors} = \sqrt{n}$, $\text{min\_dist} = 0.1$
Таким образом, алгоритм UMAP
- Обладает высокой скоростью работы на больших наборах данных
- Сохраняет глобальную структуру
- Устойчив к разной плотности (что видно на примере выше)
- Более универсален, в отличии от t-SNE
Также стоит учесть недостатки:
- Чувствительность к параметрам, результаты сильно зависят от выбора
n_neighbors - Случайность
- Сложность интерпретации расстояний